![[Logo CIB]](/imagenes/logo_cib.jpg) CENTRO DE INVESTIGACIONES BIOLÓGICAS
CENTRO DE INVESTIGACIONES BIOLÓGICASDepartamento de Biología Celular y Desarrollo
Biología Molecular de los Cromosomas
Logros más relevantes
![[Logo CIB]](/imagenes/logo_cib.jpg) CENTRO DE INVESTIGACIONES BIOLÓGICAS
CENTRO DE INVESTIGACIONES BIOLÓGICAS
Departamento de Biología Celular y Desarrollo
Biología Molecular de los Cromosomas
Logros más relevantes
![]()
Logros más relevantes
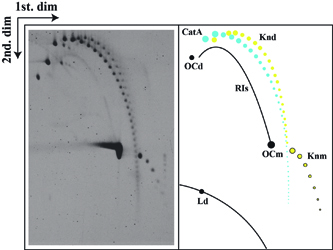 |
Autoradiografía de un gel bidimensional correspondiente a pBR18 aislado de células de E. coli de la estirpe DH5alphaF' después de su exposición a norfloxacina y digestión completa con la endonucleasa de restricción de cadena sencilla Nb-BsmI. Un diagrama iterpretativo de la autoradiografía se muestra a la derecha. En el mismo los encadenados de tipo A (CatAs) aparecen en azul claro. RIs=intermediarios de replicación con una rotura de cadena sencilla; Knd=moléculas anudadas con roturas de cadena sencilla; OCd=dimeros relajados; OCm=monómeros relajados; Knm=monómeros anudados con roturas de cadena sencilla; Ld=dímeros lineales; Lm=monómeros lineales. (Martínez-Robles et al., 2009). |
 |
Diagrama que ilustra los cambios que ocurren en la topología de un intermediario de replicación (RI) causados por el regreso de una horquilla. Se muestran paso a paso los cambios inducidos por un tratamiento con una solución 0.1 M de NaCl a 65ºC en un RI con una rotura de cadena sencilla. (A) RI parcialmente replicado con una rotura de cadena sencilla en la región no replicada. (B) El regreso de una horquilla da lugar a la formación de un duplex de DNA formado por las dos cadenas de reciente síntesis. (C) A medida que el regreso de la horquilla progresa, aumenta la longitud del cuarto brazo (rojo-rojo) y la correspondiente reducción del tamaño de la burbuja replicativa. (D) La extrusión completa del cuarto brazo da lugar a dos moléculas independientes: una molécula circular idéntica a la molécula no replicada con una rotura de cadena sencilla (OC) y otra molécula linear. Las cadenas parentales se indican en color azul y verde y las cadenas nacientes en color rojo (Fierro-Fernández et al., 2007). |
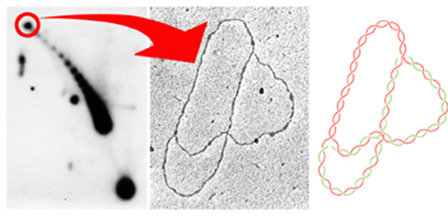 |
Para
estudiar la topología
de plásmidos parcialmente replicados, construimos una
serie de derivados de pBR322 con un terminador polar de la replicación
(TerE) a una distancia variable del origen ColE1. La autoradiografía
de la izquierda corresponde al análisis por electroforesis
bidimensional en geles de agarosa del plásmido pBR18-TerE@EcoRI
intacto. El círculo rojo señala las formas relajadas
del plásmido parcialmente replicado (OCRIs). Estas moléculas
fueron aisladas de geles bidimensionales y estudiadas al microscopio
electrónico tal como indica la flecha. La imagen del centro
corresponde a una fotografía al microscopio electrónico
de una de estas moléculas (Microscopía electrónica:
José Manuel Sogo, Zürich, Suiza). El esquema de la
derecha representa la molécula fotografiada en la que
las cadenas de DNA parentales aparecen en color rojo y las cadenas
nacientes en color verde (Esquema: Andrzej Stasiak, Basilea,
Suiza). El brazo no replicado corresponde al 43% del plásmido
en tanto que cada uno de los dos brazos ya replicados corresponde
al 57% (Olavarrieta et
al., 2002). |
 |
El análisis por electroforesis
bidimensional en geles de agarosa y microscopía electrónica
de intermediarios de replicación de plásmidos bacterianos
con la horquilla de replicación detenida nos ha permitido comprobar
que las dobles hélices hermanas pueden presentar nudos
en la región ya replicada detrás de la horquilla.
Los cruces de estas burbujas de replicación anudadas ("Knotted
Bubbles") son mayoritariamente de signo positivo. |
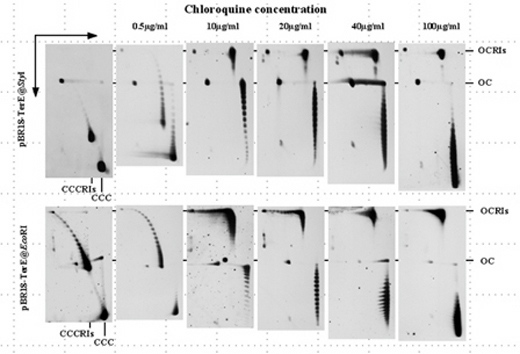
El análisis por electroforesis
bidimensional en geles de agarosa de plásmidos con la
horquilla detenida (pBR18-TerE@StyI y pBR18-TerE@EcoRI)
nos ha permitido comprobar que a diferencia de lo que ocurre
con las formas no replicadas (CCC), la cloroquina es incapaz
de inducir el superenrollamiento positivo de las formas parcialmente
replicadas (CCCRIs). Esto se debe a que en moléculas con
una horquilla detenida el superenrollamiento positivo es adsorbido
por una regresión de las horquillas con la consiguiente
formación de estructuras de tipo Holliday (Olavarrieta
et al., 2002). |
 |
 |
La
muestra (pHH5.8) fue analizada por electroforesis bidimensional en un
gel de agarosa luego de su digestión con AlwNI (ángulo
superior izquierdo), enriquecida en los intermediarios indicados por el
origen de la flecha, el DNA recubierto con la proteína
recA y examinada al microscopio electrónico (Sogo
et al., 1999). En el ángulo inferior derecho se muestra
un detalle a mayor aumento de la región donde se cruzan
las dos dobles hélices hermanas en el que se puede apreciar
en cada cruce cuál de los segmentos pasa por arriba y
cuál por abajo. (Microscopía electrónica:
José Manuel Sogo, Zürich, Suiza). |
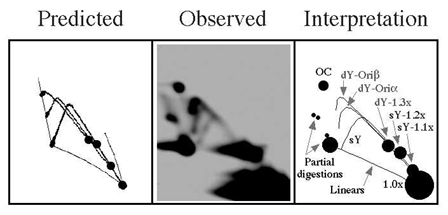
Una
de las técnicas
que utilizamos en el laboratorio es la "Electroforesis bidimensional
en geles de agarosa" (2D gels). Esta técnica fue
desarrollada originalmente por Bonita Brewer y Walton Fangman
(Seattle, Washington, EEUU) para averiguar la forma de los intermediarios
de replicación de cualquier fragmento de DNA linear. Con
el fin de ayudar a todos aquellos interesados en conocer mejor
y utilizar esta técnica, hemos desarrollado un programa
informático que permite predecir los patrones generados
por los intermediarios de replicación de cualquier fragmento
linear analizado por electroforesis bidimensional en geles de
agarosa (Viguera et al., 1998).
Si quieres copiar el programa simulador "2D gel", selecciona aquí. |
 |
Autor: Jorge Bernardo Schvartzman Blinder
Madrid, Noviembre 2003Docmaster: J.R. Díez
Madrid, 28 de Septiembre de 1998
ir a:
Página principal del Laboratorio de Biología Molecular de los Cromosomas
Página principal del Departamento de Biología Celular y del Desarrollo
Página principal del Centro de Investigaciones Biológicas